Sage Science核酸片段回收系統(tǒng)在RADseq中的應(yīng)用
1.什么是RAD-SEQ?
RADseq(Restriction-site associated DNA sequencing,限制性酶切位點相關(guān)的DNA測序)是通過對基因組進(jìn)行酶切,并針對帶有酶切位點的DNA相關(guān)片段進(jìn)行測序,這項技術(shù)能夠低成本的開發(fā)大量SNP標(biāo)記,不受有無參考基因組和染色體倍型的限制。
2.它的應(yīng)用在哪方面?
RADseq技術(shù)在動植物的群體進(jìn)化研究,遺傳圖譜構(gòu)建和QTL定位,以及全基因組關(guān)聯(lián)分析上得到了廣泛的應(yīng)用。其優(yōu)勢應(yīng)用是非模式物種的生態(tài)與進(jìn)化研究中的SNP查找與基因分型研究。
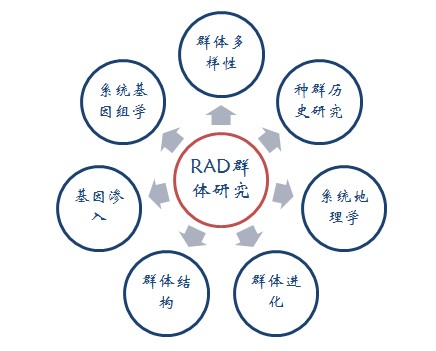
3.RAD-seq的種類?
1) RestSeq(restriction fragment sequencing):限制性酶I酶切+連接接頭+限制性酶II酶切+片段選擇建庫測序;
2) ezRAD:一個或多個常見酶酶切+illuminate建庫Kit建庫測序;
3) SLAF-seq(specific-lo­cus amplified fragment sequencing):酶I酶切+barcode+酶II酶切+接頭建庫測序;
4) 2bRAD : IIB限制性酶單酶切,IIB型限制性內(nèi)切酶能在識別位點的上游和下游位點分別切斷,獲得固定長度的片段。構(gòu)成33–36 bp插入片段建庫測序;
5) ddRAD(double-digest restriction-site-associated DNA):兩個限制性酶酶切,接頭分別與一種酶配套+切膠進(jìn)行片段選擇建庫測序;
6) SBG(sequence-based genotyping):稀有酶和一個或兩個常見酶酶切+PCR選擇性擴(kuò)增短片段序列建庫測序;
7) MSG(multiplexed shotgun genotyp­ing):常見酶單酶切+片段選擇建庫測序;
8) GBS(genotyping by sequencing):常見酶單酶切+PCR選擇性擴(kuò)增短片段序列建庫測序;
9) RRL(reduced representation library):平性末端酶酶切+片段選擇建庫測序;
10) original RAD(original restriction-site associat­ed-DNA):限制性酶單酶切+機(jī)械打斷建庫測序;
11) CRoPS(complexity reduction of polymorphic sequences):兩個酶酶切+AFLP產(chǎn)物建庫測序;
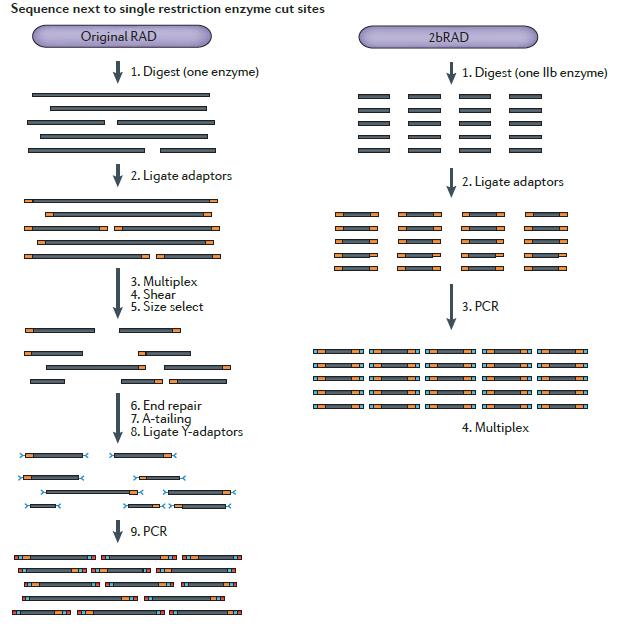
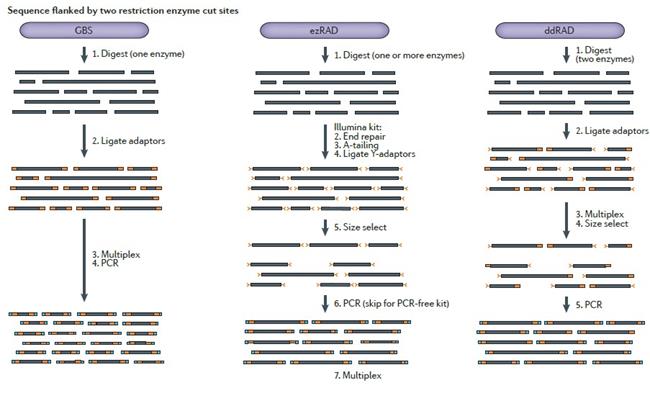
4.RAD-seq 文庫構(gòu)建過程中,DNA片段大小篩選的重要意義?
酶切可以將基因組簡化為很多DNA片段。而片段大小篩選步驟可以進(jìn)一步減少需要進(jìn)行基因分型的基因位點。篩選潛在的特征性基因座位,主要依賴于兩酶切位點距離。不同測序文庫間片段篩選的一致性,對產(chǎn)生不同樣本間可對比的基因座位至關(guān)重要,進(jìn)而直接影響后續(xù)的生物信息分析和應(yīng)用。片段篩選的方式有直接篩選和間接篩選。直接篩選包括手工切膠,磁珠篩選和pippin,但不同的技術(shù)手段,其精確性不同。下圖為不同技術(shù)手段的簡單比較。

手工切膠是根據(jù)最左邊的marker對照切割,即使是同一個人操作,也很難保證每個泳道,每個樣品目的范圍的一致性。
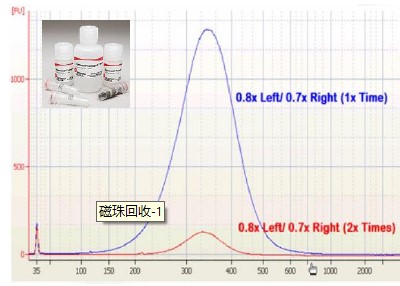
磁珠純化的片段范圍過于寬泛,無法進(jìn)行聚焦。
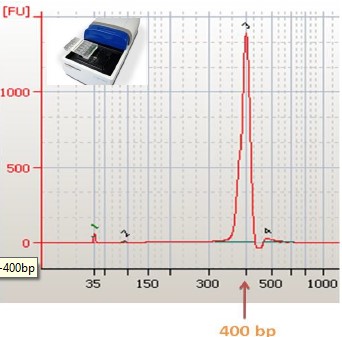
相比于手工切膠和磁珠純化,pippin自動化片段回收的一致性和準(zhǔn)確性更高,重復(fù)性和精確度均達(dá)到95%以上。在ezRAD,ddRAD文庫構(gòu)建過程中,pippin片段回收的一致性,是實驗成功的關(guān)鍵因素,ddRAD的創(chuàng)始人Brant Peterson博士認(rèn)為“即使在手工切膠最佳的實驗案例里,生信分析后發(fā)現(xiàn),手工切膠的效果也僅為pippin儀器的一半”。自2011年第一代產(chǎn)品Pippin Prep發(fā)布以來,已陸續(xù)推出Blue Pippin、SageELF、PippinHT、SageHLS等多個型號,sage science在核酸片段回收領(lǐng)域已成為領(lǐng)導(dǎo)性品牌。
環(huán)亞生物科技有限公司是美國Sage公司中國區(qū)總代理商,一直關(guān)注測序相關(guān)設(shè)備在國內(nèi)的引進(jìn)和開發(fā),致力于協(xié)助國內(nèi)客戶更好、更快完成測序樣本的制備工作,實現(xiàn)更高效率、更輕松的工作流程。
公司網(wǎng)站:www.apgbio.com
參考文獻(xiàn):
Andrews K R, Good J M, Miller M R, et al. Harnessing thepower of RADseq for ecological and evolutionary genomics.[J]. Nature ReviewsGenetics, 2016
Peterson, B. K., Weber, J. N., Kay, E. H., Fisher, et al. Double digest RADseq: an inexpensive method for de novo SNP discovery and genotyping in model and non-model species.[J]. PLOS ONE , 2012
標(biāo)簽:
RAD-SEQ 限制性酶切位點
Copyright(C) 1998-2024 生物器材網(wǎng) 電話:021-64166852;13621656896 E-mail:[email protected]