精確修飾位點譜圖庫的建立與磷酸化蛋白質(zhì)組的 DIA 解析
張偉 周岳
賽默飛世爾科技(中國)有限公司
關鍵詞
翻譯后修飾;DIA;譜圖庫;磷酸化;定量蛋白質(zhì)組學
引言
數(shù)據(jù)非依賴采集(Data-Independent Acquisition, DIA)是當前最熱門的質(zhì)譜采集技術之一,它以非目標的方式將質(zhì)量范圍分為若干窗口,依次并循環(huán)采集窗口內(nèi)所有母離子的二級碎片[1,2]。DIA 與 SRM 類似,也是基于子離子(transition)定量,相比傳統(tǒng)蛋白質(zhì)組學定量方法具有更好的選擇性和更高的準確度。然而,目前DIA在翻譯后修飾分析上仍有較大瓶頸。DIA 依賴于 DDA 建立譜圖庫,而 DDA 數(shù)據(jù)在搜庫鑒定時,修飾位點定位錯誤的概率較高,特別是磷酸化修飾發(fā)生在常見的 S/T/Y 上,若肽段含有 2 個或以上位置接近的 S/T/Y,位點就容易找錯。將含有錯誤位點信息的鑒定結果作為譜圖庫,就會導致翻譯后修飾 DIA 解析結果的不可靠[3]。因此,DIA 尚難用于大規(guī)模的翻譯后修飾樣本分析。
針對修飾位點的打分算法使修飾位點的定位更加準確,Proteome Discoverer 軟件中整合的 phosphoRS/ptmRS 模塊[4]和 MaxQuant 軟件的算法[5]都可以實現(xiàn)位點可信度(Site Probability)的計算,從而獲得可靠的位點定位信息。本文基于上述軟件對翻譯后修飾 DDA 數(shù)據(jù)進行位點可信度分析,篩選具有準確位點定位的譜圖建立譜圖庫,導入 Skyline 軟件,進而實現(xiàn)可靠的翻譯后修飾 DIA 解析。將該流程應用于磷酸化樣本 DIA 數(shù)據(jù)分析,成功提取 6401 條高可信度的磷酸化肽段(Q < 0.01),占譜圖庫肽段總數(shù)的 98.4%,其中可用于準確定量的肽段(CV < 20%)占 86.9%,有效解決了翻譯后修飾 DIA 定量的難題。
實驗條件
實驗材料和方法
來源于大鼠組織富集的磷酸化樣本,最終上樣量為 700 ng/run,分別進行 DDA 和 DIA 采集,每種采集模式重復 3 遍。
色譜條件
納流高效液相色譜儀:EASY-nLC 1000(Thermo Scientific)
分析柱:納流 C18 色譜柱(長15cm, ID 75 μm, 粒徑3 μm)
流動相:A:0.1% 甲酸水溶液;B:0.1% 甲酸乙腈溶液
梯度:0–3 min, 3–7% B; 3–95 min, 7–22% B; 95–113 min, 22–35% B; 113–116 min, 35-90% B; 116-120 min, 90% B
流速:300 nL/min
質(zhì)譜條件
質(zhì)譜儀:Orbitrap Fusion(Thermo Scientific);
離子源:NanoFlex;離子模式:正離子;噴霧電壓:1.8 kV;毛細管溫度:275°C;S-Lens RF:60%
DDA:分辨率:一級120,000@m/z 200,二級30,000@m/z 200;AGC:一級2e5,二級5e4;二級Maximum Injection Time:100 ms;碰撞能量:HCD 30%
DIA:質(zhì)量范圍:m/z 400–1200;窗口:25 m/z(窗口間 1 m/z 重疊,實際 Isolation window 設 26 m/z);二級分辨率:30,000@m/z 200;二級AGC:1e5;二級 Maximum Injection Time:85 ms;碰撞能量:HCD 30%;每個 DIA 循環(huán)之間插入一次一級掃描。
數(shù)據(jù)處理
基于 Proteome Discoverer 2.0 軟件進行 DDA 鑒定、位點篩選和譜圖庫建立,Proteome Discoverer 1.4 和 MaxQuant 也可以實現(xiàn)相同的工作。
搜庫鑒定參數(shù):Uniprot 大鼠蛋白數(shù)據(jù)庫,母離子質(zhì)量偏差:10 ppm;碎片離子質(zhì)量偏差:0.02 Da;固定修飾:C 烷基化(+57.021 Da);動態(tài)修飾:M 氧化(M+15.995 Da);S/T/Y 磷酸化(S/T/Y+79.966 Da);酶:trypsin;Q 值(Percolator):< 0.01;ptmRS 模塊:PhosphoRS mode: True; Use diagnostic ions: True
位點篩選和譜圖庫建立:對 PSM 表格“Isoform Confidence Probability”一項進行篩選,保留 _ 0.75 的結果,導成 PepXML 格式,并將相應譜圖導成 mzML 格式。PepXML 文件和 mzML 文件同時導入 skyline 建立高可信磷酸化肽段譜圖庫。
DIA 數(shù)據(jù) Skyline 解析:根據(jù) DIA 采集參數(shù)設置隔離窗口,將譜圖庫中所有肽段及相應蛋白作為 Targets,每個肽段選取強度最高的 6 個 b/y 離子進行峰抽提,提峰結果使用 mProphet 進行假陽性評估,控制 Q 值 < 0.01。
實驗結果
1. 精確修飾位點譜圖庫建立與 DIA 解析流程
由于肽段中含有多個可能發(fā)生翻譯后修飾的位點,如果不對可能發(fā)生修飾的位點進行可信度打分,會造成翻譯后修飾位點的錯誤匹配和定位。將錯誤匹配和定位的翻譯后修飾肽段作為譜圖庫,就會導致 DIA 解析結果的錯誤。這是目前翻譯后修飾 DIA 分析的瓶頸所在。
基于 Proteome Discoverer 的 phosphoRS/ptmRS 算法能夠?qū)赡馨l(fā)生修飾的位點進行打分(Site Probability),判斷定位的準確性。通常認為 Probability _ 75(100 分制)或 0.75(1 分制)的位點定位準確、可靠;具有多個修飾位點的肽段,其所有位點的 Probability 均 _ 75(或 0.75),則修飾位點及 isoform 唯一確定。通過這一方法,篩選出位點準確可信、isoform 唯一確定的 PSM(即 Isoform Confidence Probability 75(或0.75)建立譜圖庫,實現(xiàn)精確的翻譯后修飾 DIA 分析。整個流程如圖 1 所示。
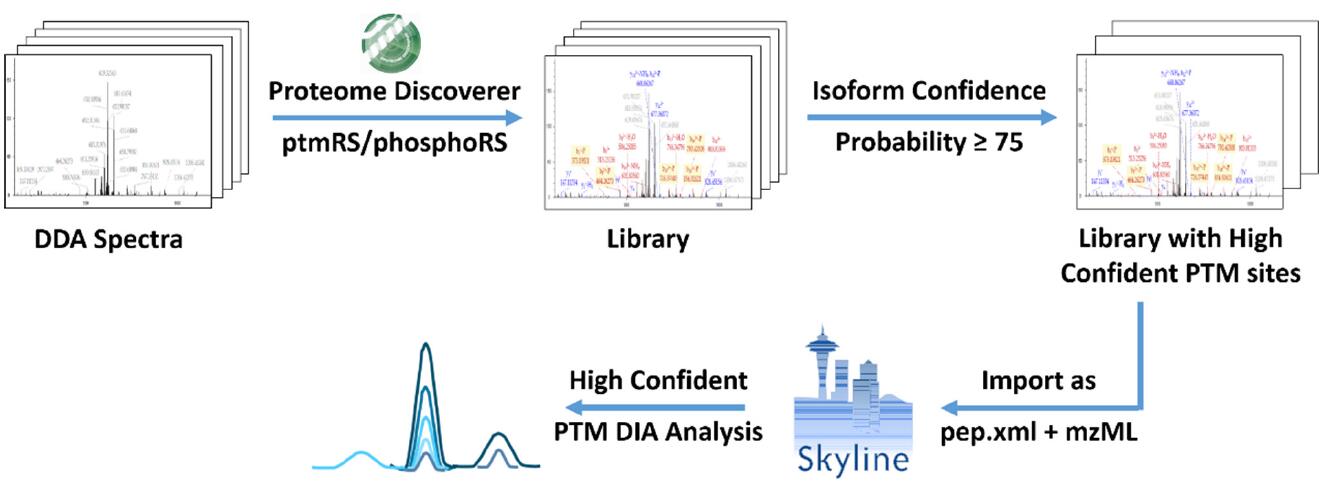
圖 1. 精確修飾位點譜圖庫建立流程圖
2. 磷酸化樣本的 DDA 鑒定、可信度篩選和譜圖庫建立
磷酸化樣本信息和色譜質(zhì)譜參數(shù)見實驗條件部分。3 針 DDA 數(shù)據(jù)按磷酸化檢索流程使用 Proteome Discoverer 2.0 軟件搜庫鑒定(S/T/Y+79.966 Da),并使用 ptmRS 模塊對位點打分(圖 2-1)。搜庫完成后,打開結果,使用 Filter 功能對 PSM 列表“Isoform Confidence Probability”項進行篩選,保留得分大于等于 0.75 的 PSM(圖 2-2)。然后,點右鍵選擇”Check All-In This Table”,將符合條件的PSM選定。最后,在”Spectra”中導出mzML格式,在“To PepXML”中導出 PepXML 格式,并將兩個文件置于同一文件夾中,即可作為譜圖庫導入 Skyline(圖 2-3)。
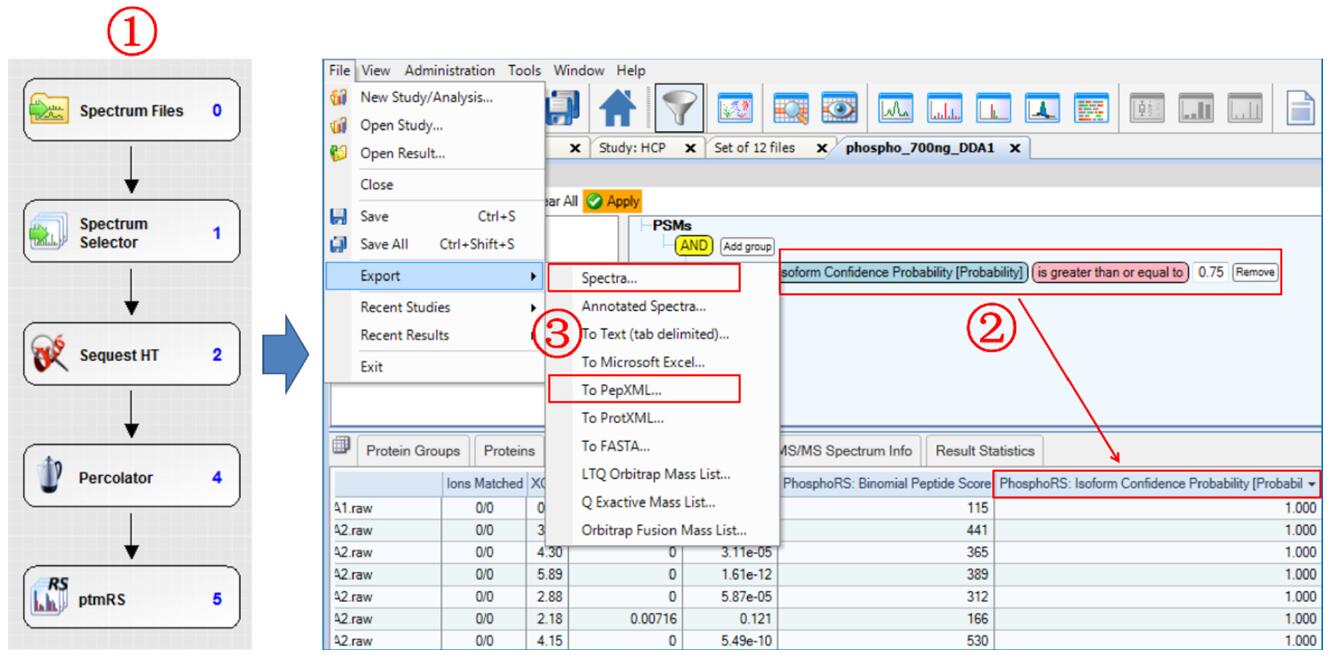
圖 2. 翻譯后修飾 DDA 數(shù)據(jù)檢索、位點篩選和結果導出步驟
經(jīng) phosphoRS/ptmRS 計算得到每個磷酸化位點的打分(Site Probability),分數(shù) > 75(或 0.75)則位點有確切的碎片支持,定位可靠。搜庫引擎沒有針對位點打分的功能,位點定位錯誤概率大。圖 3 是一個典型的例子:RFSVTAEGGLTLEQVTDAR 肽段搜庫鑒定出包含 1 個磷酸化位點的多個 PSM,3 號位絲氨酸和 5 號位蘇氨酸均有匹配到磷酸化發(fā)生(FDR < 1%)。而 ptmRS 的打分結果卻分 3 種情況:1) ptmRS 得到唯一可靠的位點,且與搜索引擎得到的位點一致,此時 Isoform 100% 確定(圖 3-A);2) ptmRS 得到兩個位點都有可能,無法確定唯一位點,此時 Isoform 可靠度為 50%(圖 3-B);3) ptmRS 得到唯一可靠的位點,但與搜索引擎得到的位點不一致,此時 Isoform 可靠度為 0(圖 3-C)。最終,只篩選 Isoform打分(Isoform Confidence Probability)_ 75(或 0.75),即所有位點均明確、可靠的肽段建立譜圖庫。
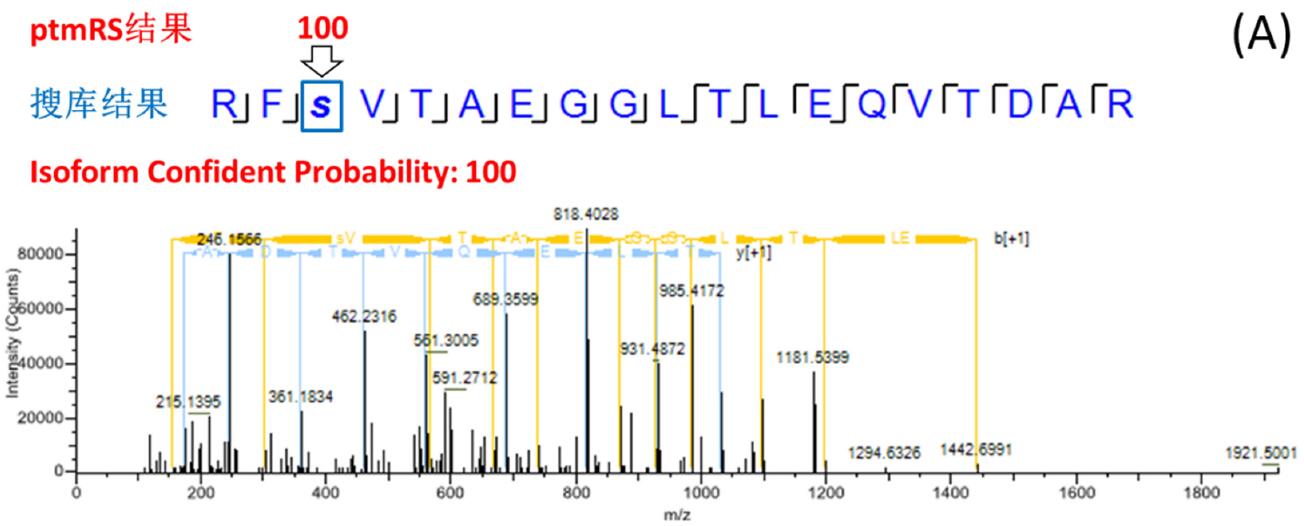

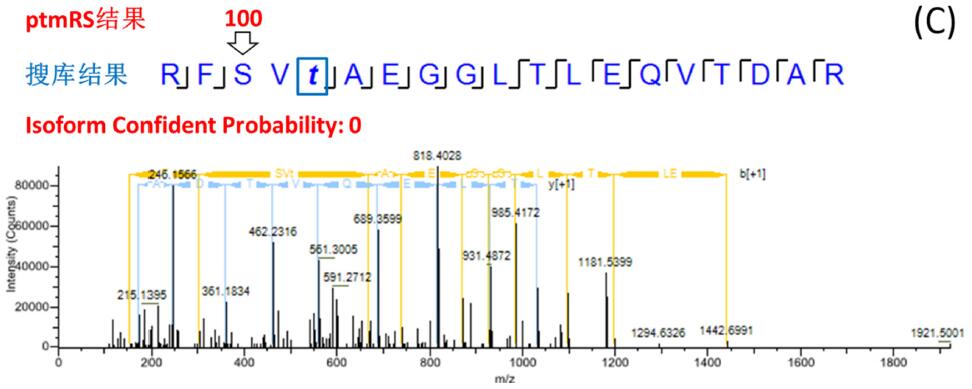
圖3. 搜庫引擎/ptmRS結果一致、部分一致、不一致三種情況示例
實驗共鑒定 56617 張 PSM(FDR < 1%),經(jīng)過 Isoform Confident Probability 篩選,共獲得 30558 張精確定位、isoform 唯一的磷酸化肽 PSM(圖 4),結果導成 mzML 和 PepXML 格式作為譜圖庫。
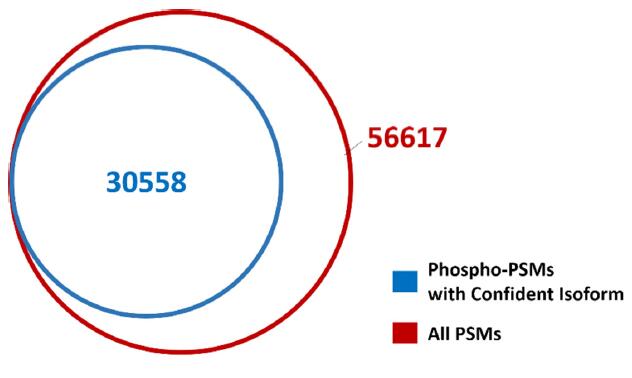
圖 4. Isoform Confident Probability 篩選前后 PSM 比較
3. 基于精確譜圖庫的磷酸化樣本 DIA 數(shù)據(jù)解析
將 mzML 和 PepXML 格式文件導入 Skyline 生成磷酸化肽譜圖庫,用于磷酸化肽段 DIA 數(shù)據(jù)解析。Skyline 將譜圖庫中所有肽段作為 targets 對 DIA 數(shù)據(jù)中進行提峰,每條肽段選取 3 個母離子(單同位素, 單同位素 +1, 單同位素 +2)和 6 個響應最高的 b/y 離子。提峰結果使用 mProphet 進行假陽性評估,Q 值 < 0.01(即 FDR < 1%)為可信的提峰結果。
結果顯示,從 DIA 數(shù)據(jù)中提取、定量到 6401 條可信的磷酸化肽,占譜圖庫磷酸化肽總數(shù)(6505 條)的 98.4%(圖 5)。磷酸化肽的豐度和離子化效率普遍較低,本實驗如此高的解析成功率表明,基于 Orbitrap 的 DIA 數(shù)據(jù)具有極高的譜圖質(zhì)量和出色的靈敏度。
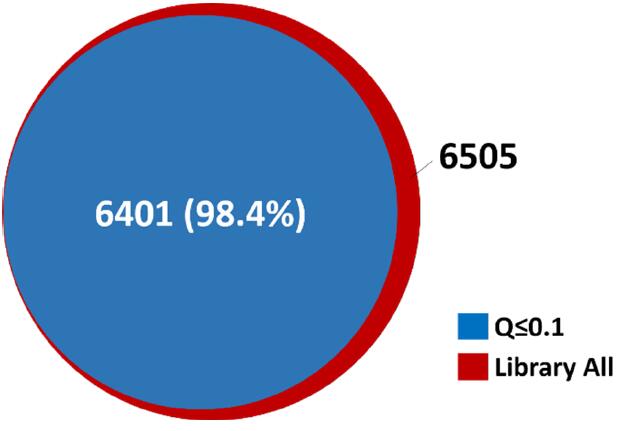
圖5. DIA可靠解析的磷酸化肽占譜圖庫總數(shù)的98.4%
進一步對 6401 條磷酸化肽 XIC 色譜峰面積的三針重現(xiàn)性進行統(tǒng)計。將每條肽的母離子和子離子峰面積分別加和,選取兩者中重現(xiàn)性最好(即 CV 值最低)的結果用于定量。一般認為,峰面積 CV 值 < 20% 時定量結果可靠、準確。統(tǒng)計結果顯示,峰面積 CV 值 < 20% 的磷酸化肽占總數(shù)的 86.9%(圖 6)。如此高的重現(xiàn)性說明 Orbitrap DIA 不僅具有出色的靈敏度,同時具有優(yōu)越的重現(xiàn)性,勝任復雜的翻譯后修飾定量。

圖 6. 磷酸化肽峰面積重現(xiàn)性(CV 值)統(tǒng)計
圖 7 展示了一個典型的磷酸化肽 DIA 解析結果。該磷酸化肽有 2 種異構體,分別在 3 號位和 9 號位的絲氨酸上發(fā)生了磷酸化。得益于高質(zhì)量的 DIA 譜圖和嚴格的譜圖庫建立,即使這 2 種異構體保留時間非常接近,也能成功、準確地分辨。DIA 獲得的碎片豐度比與譜圖庫非常接近,匹配打分的 dotp 值均在 0.95 以上。

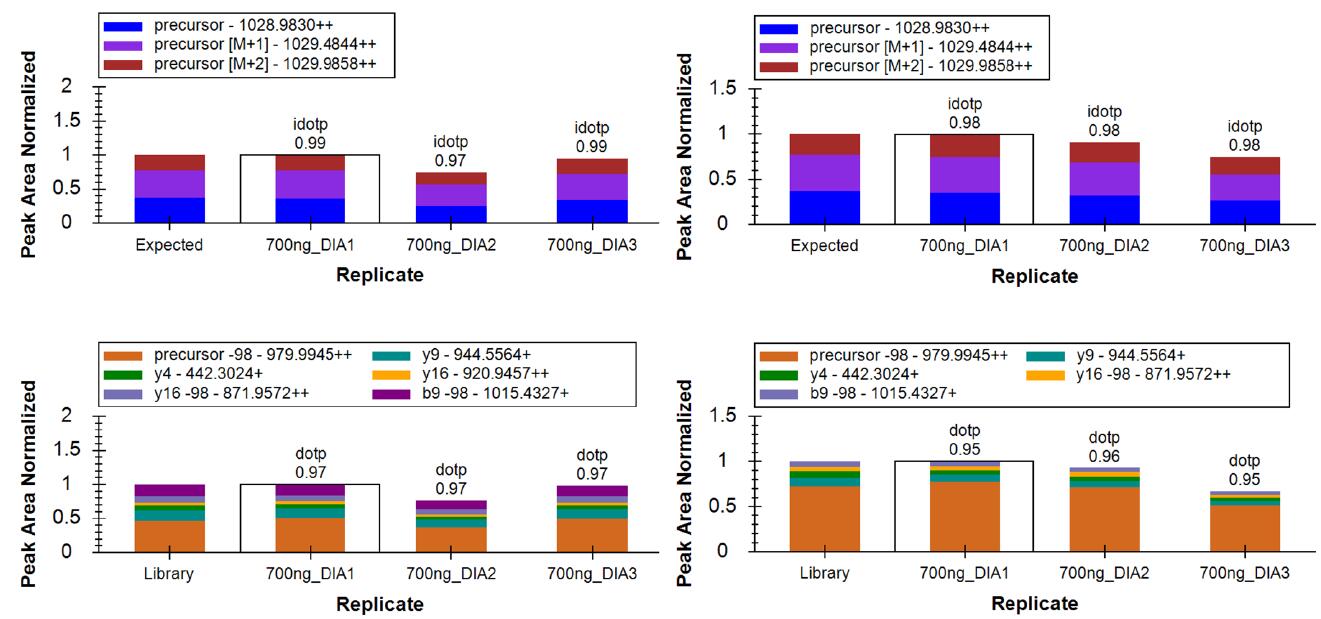
圖 7. 磷酸化肽異構體在 DIA 中的分辨和匹配打分(dotp)
結論
翻譯后修飾由于肽段上修飾位點的多重性和不確定性,難以獲得可靠的 DIA 結果,這一問題是 DIA 發(fā)展的瓶頸所在。另一方面,phosphoRS/ptmRS 和 MaxQuant 等算法和軟件的發(fā)展,使翻譯后修飾位點的 DDA 鑒定、定位更加準確。本文基于上述算法和軟件對磷酸化樣本的 DDA 鑒定結果進行位點打分,并篩選出位點定位準確、可靠的譜圖建立譜圖庫用于 DIA 分析,成功從磷酸化樣本 DIA 數(shù)據(jù)中提取 6401 條可靠的磷酸化肽(Q < 0.01),占譜圖庫中磷酸化肽總數(shù)的 98.4%。其中,86.9% 的肽段峰面積 CV < 20%,可用于精確的磷酸化定量。該策略有效解決了翻譯后修飾 DIA 定量的難題,證明基于 Orbitrap 超高分辨質(zhì)譜技術的 DIA 流程兼具出色的靈敏度和優(yōu)越的重現(xiàn)性,是復雜樣本定量特別是翻譯后修飾樣本定量的最佳選擇。
參考文獻
[1] Multiplexed peptide analysis using data-independent acquisition and Skyline, Nat. Protoc., 2015, 10(6): 887-903
[2] Extending the limits of quantitative proteome profiling with data-independent acquisition and application to acetaminophen-treated three-dimensional liver microtissues, Mol. Cell. Proteomics, 2015, 14(5): 1400-1410
[3] MS1 Peptide Ion Intensity Chromatograms in MS2 (SWATH) Data Independent Acquisitions. Improving Post Acquisition Analysis of Proteomic Experiments, Mol. Cell. Proteomics, 2015, 14(9): 2405-2419
[4] Universal and confident phosphorylation site localization using phosphoRS, J. Proteome Res., 2011, 10(12): 5354-5362
[5] MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification, Nat. Biotechnol., 2008, 26(12): 1367-1372
Copyright(C) 1998-2024 生物器材網(wǎng) 電話:021-64166852;13621656896 E-mail:[email protected]